環形細菌染色體
環形細菌染色體是指呈環形DNA分子的細菌染色體。和大多數真核生物的線性DNA不同,典型的細菌染色體是環形的。
大多數細菌的染色體組由環形的DNA分子構成,這種DNA是沒有自由端的。自由端將會給DNA的複製及其穩定性帶來巨大困難。含有帶DNA端或端粒(大多數真核生物)的染色體的細胞,進化演變出了複雜的機制來克服這些困難。 然而,環形的染色體也給細胞帶來了其他的挑戰。複製後,兩個子代環形染色體有時可能會依然相互連接着或糾纏在一起,必須解決這些問題才能使得在細胞分裂時兩個子代細胞均能獲得完整的染色體副本。
環形細菌染色體的複製[編輯]
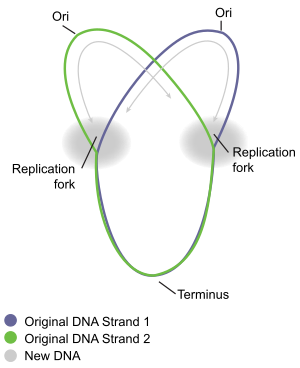
要理解原核生物DNA複製的過程,最好是藉助已被充分研究的大腸埃希氏菌和枯草芽孢桿菌。染色體複製過程由三個主要階段組成:初始階段、延伸階段和終止階段。染色體起始位置稱為複製起點(oriC),在此處有序地合成「啟動器」蛋白是初始階段的開始。該蛋白的合成過程經過調控,確保在每個細胞周期只會發生一次染色體複製。初始階段中複製起點會合成一種酶,在延伸階段時,兩條臂上的酶朝着相反的方向複製DNA,最終生成兩條一模一樣的染色體。這一過程被稱為雙向複製。每根染色體臂上參與DNA複製過程的整個分子結構叫做「複製體」。在複製體最前端的是解旋酶,它解開兩條螺旋在一起的DNA鏈,形成一個移動的「複製叉」。兩條被解開的DNA單鏈用作DNA聚合酶的複製模板。每條DNA單鏈上聚合酶與解旋酶(連同其他的蛋白質)一起移動,合成一條DNA副本補鏈。如此進展下去,產生了兩條與原DNA鏈一模一樣的副本鏈。最終,兩個複製叉沿着環形染色體再次相遇,相遇的位置大致與複製起點相對,稱為終止位置。隨後延伸酶分解。在細胞分裂完成之前,形成兩個子代染色體。
初始階段[編輯]
大腸桿菌的複製起點包含能夠被DnaA蛋白質識別的核酸序列,不同種類的細菌都會將自身的該序列嚴密保護起來。複製起點的DnaA蛋白在調控之下召集其他酶和蛋白質,形成兩個完整的複製體來進行雙向複製。[1]
複製起點內的DNA序列信息對其功能十分重要。其中包括DnaA箱,它是一段重複的9鹼基序列:5'-TTATCCACA-3'[2],可被DnaA蛋白識別。 DnaA蛋白在染色體DNA複製中發揮着重要作用。[3] 結合ATP,在類組織蛋白[HU]的協助下,DnaA蛋白在複製起點的左側解開一個富三腺苷區域,它將會攜帶13段結構基元[4],並將打開這兩股DNA鏈以便其他複製蛋白進去該區域。[5]
該區域包含四個「GATC」序列,能夠被DNA腺甲基化酶 (DAM)識別。還包含一種能夠在序列未甲基化或者半甲基化的時候修改腺嘌呤的酶。腺嘌呤的甲基化十分重要,因為它將改變DNA的構造以促進DNA鏈的分離[6]。複製起點的這一區域,有解旋的自然傾向。[7]
接下來DnaA蛋白會從DnaB-DnaC綜合體中召集解旋酶,既DnaB,到被解旋的區域來形成預啟動綜合體[8]。在DnaB移位到每個複製叉頂部後,解旋酶解開兩條DNA父鏈,立刻和引發酶發生反應。[9]
為了DNA複製的順利進行,需要單鏈的綁定蛋白質來防止DNA單鏈形成次生結構及二次復性。此外,需要DNA旋轉酶來釋放由DnaB解旋酶作用造成的拓撲結構上的作用力。
延伸階段[編輯]
當複製叉沿着染色體環移動時,形成了一個形似希臘字母西塔Ө的結構。英國生化學家約翰·凱恩斯通過一種創新的方式將DNA複製可視化,在1963年提出了大腸桿菌染色體複製過程中的西塔結構。在他的實驗中,他使用含有3H-胸腺的培養液來培養培養基以對染色體進行放射性標記。被標記的核苷基團均勻地混入到細菌染色體中。接下來他小心地將細胞溶解並放到電子顯微鏡(EM)網格下,通過兩個月的X射線照射來分離出染色體。這一實驗清晰地闡釋了環形細菌染色體的西塔複製模型。[10]
如上所述,細菌染色體的複製以雙向的方式進行。通過對複製中的細菌染色體進行放射性同位素標記,第一次展示了這種複製方式。後來試驗中正在進行複製的DNA區域通過放射能圖像技術被可視化,洗出的膠片可在顯微鏡下進行細緻地觀察。這使得研究人員能夠看到複製發生的位置。關於雙向複製的首次結論性觀察來自於對枯草芽孢桿菌研究[11]。不久之後,觀察發現大腸桿菌染色體也是雙向複製的。[12]
- 參見D.M.Prescott和P.L.Kuempel的1972年論文中的圖4:細胞中經19分鐘[3H]胸腺嘧啶標記,及2.5分鐘[3H]胸腺嘧啶標記和[H]胸苷標記後的大腸桿菌染色體所產生的紋理軌跡。
大腸桿菌的DNA聚合酶III全酶是一種900 kD綜合體,本質上呈聚合結構。每個單體包含一個催化核、一個二聚作用的亞單元和一個具有持續合成能力的亞單元。[13] DNA聚合酶III利用自身的一個核心亞單元,來不斷地合成前導鏈,而另一套核心亞單元在環形的後隨鏈上從一個岡崎片段移動到下一個岡崎片段。前導鏈的合成開始於一小段RNA引物的合成,發生在複製起點,由引發酶(DnaG蛋白質)催化進行。
隨後在DNA聚合酶III聚合體和DnaB解旋酶的聯合作用下,去氧核苷酸被添加到這段引物上。之後連續地進行前導鏈的合成,此時在複製叉處的DNA處於解旋狀態。與此不同的是,後隨鏈的合成通過一個個的岡崎片段完成。首先,引發酶合成一段RNA引物;隨後,與前導鏈的合成類似,由DNA聚合酶III與RNA引物結合併向後隨鏈添加去氧核糖核苷酸。
當一個岡崎片段合成完畢時,複製停止。DNA聚合酶III的核心亞單元與β滑動夾分離[β滑動夾是DNA聚合酶III的具有持續合成能力的亞單元][14]。在DNA聚合酶I[它還對外切酶的活動進行校對]的作用下,RNA引物被移除,由DNA片段取而代之。隨後,DNA連接酶將缺口封住,並將這些片段連接起來形成後隨鏈。
終止階段[編輯]
DNA複製終止是指複製叉融合及複製體解體,以產出兩個獨立且完整的DNA分子的過程。它發生在複製終點區域,在染色體上的位置大致與複製起點相對(圖5)。終點區域包含多個DNA複製終結站,稱為Ter站。需有一個特殊的「複製終結者」蛋白綁定在Ter站才能是複製過程停下來。每個Ter站都有活動極性,也即是說,它將使從某一個方向靠近Ter站的複製叉停止,而從另一個方向靠近過來的複製叉活動不受影響。多個Ter站排列形成兩個相對的小組,以使兩個方向來的複製叉在他們相遇的這一區域內停下來。這種排列被稱為「複製叉陷阱」。[15]
- 參見大腸桿菌複製Ter站的位置和順序:(A)複製起點和10個複製Ter站位置的圖示。(B)複製重點的一致順序。
在大腸桿菌中,Ter站專門與複製終結者蛋白Tus發生反應。[16] Tus-Ter綜合體依照方向性,阻止DnaB解旋酶的DNA解旋作用。[17]
DNA的複製將相對的複製叉分離,使完整的染色體結合成為「索烴」或拓撲結構上的相互關聯的環形。這些環形並不是共價相連,但卻不能分開,因為它們內旋在一起,並且每個環形都是共價閉合的。這些成鏈的環形需要拓撲異構酶的作用來將之分離[稱為解鏈作用, decatenation]。在大腸桿菌中,DNA Topo IV酶在成鏈染色體的分離中有着重要作用,它將一條染色體的兩條DNA鏈暫時性的打斷,並允許其他染色體從斷裂處通過。
目前,DNA旋轉酶在解鏈作用中的作用還不太明確。從定義上來講,有兩類拓撲異構酶:第一類產生短暫的DNA單鏈斷裂;第二類產生短暫的DNA雙鏈斷裂。因此,第一類拓撲異構酶一次解開一個DNA超螺旋,而第二類酶一次解開兩個超螺旋。原核生物和真核生物中的Topo I酶都是第一類拓撲異構酶。真核生物中的Topo II酶、細菌的旋轉酶和細菌的Topo IV酶都屬於第二類拓撲異構酶。
需要記住的是DNA旋轉酶事實上進行的是第二類拓撲異構酶作用。因此,它和Topo IV酶(也進行的是第二類拓撲異構酶作用)本質上是同種物質,我們認為這兩種蛋白質的功能是相似的。DNA旋轉酶的根本作用是對DNA鏈施以反向超螺旋力,從而使DNA複製時的正向超螺旋鬆弛。Topo IV酶也能使正向超螺旋鬆弛,因此,DNA旋轉酶和Topo IV酶的作用幾乎一樣:在DNA聚合酶運作之前,使正向超螺旋鬆弛,以便DNA複製過程不受拓撲結構扭力阻礙。[18]
某些科學文獻中說DNA旋轉酶是唯一負責解鏈作用的酶,引起了爭議。 在1997年,一個由Zechiedrich、Khodursky和Cozzarelli主導的實驗發現,Topo IV酶是細菌DNA複製中間過程中唯一一種發揮作用的解鏈酶。[19]在這項實驗中,當僅抑制DNA旋轉酶時,大多數索烴照常被解鏈。然而當僅抑制Topo IV酶時,解鏈作用幾乎完全無法進行。該實驗結果表明,Topo IV是活體實驗中主要的解鏈酶。雖然DNA旋轉酶也在解鏈作用中發揮一定的作用,它在互聯的染色體解鏈過程中的功能並沒有Topo IV重要。
鳴謝[編輯]
本詞條基於2007年CC by SA許可條件下墨爾本大學微生物與免疫學學院的一門課程中Imalda Devaparanam和David Tribe撰寫的文章。
參見[編輯]
參考文獻[編輯]
本文使用的材料取自Citizendium文章《環形細菌染色體複製》,由Creative Commons Attribution-ShareAlike 3.0 Unported License授權許可,而非GFDL
- ^ Jon M. Kaguni DnaA: Controlling the Initiation of Bacterial DNA Replication and More.
- ^ C Weigel, A Schmidt, B Rückert, R Lurz, and W Messer.
- ^ Hirota Y, Mordoh J and Jacob F (1970) On the process of cellular division in Escherichia coli III.
- ^ Bramhill D, Kornberg A. 1988.
- ^ Sekimizu K, Bramhill D and Kornberg A (1987) ATP activates dnaA protein in initiating replication of plasmids bearing the origin of the E.coli chromosome.
- ^ Gotoh O, Tagashira Y. 1981.
- ^ Kowalski D, Eddy MJ. 1989.
- ^ Carr KM, Kaguni JM. 2001.
- ^ Tougu K, Marians KJ. 1996.
- ^ Cairns, J.P.: Cold Spring Harbor Symposia on Quantitative Biology 28:44, 1963.
- ^ Wake, R.G. 1972.
- ^ Prescott D.M., Kuempel P.L. 1972.
- ^ O'Donnell M. , Jeruzalmi D. , Kuriyan J. Clamp loader structure predicts the architecture of DNA polymerase III holoenzyme and RFC.
- ^ Indiani C, O'Donnell M. Mechanism of the delta wrench in opening the beta sliding clamp.
- ^ Duggin IG, Wake RG, Bell SD, Hill TM. 2008.
- ^ Kamada K, Horiuchi T, Ohsumi K, Shimamoto N, Morikawa K. 1996.
- ^ Kaplan DL, Bastia D. 2009.
- ^ Chris Ullsperger and Nicholas R. Cozzarelli.
- ^ E L Zechiedrich , A B Khodursky , N R Cozzarelli.
