脱氧核糖核酸

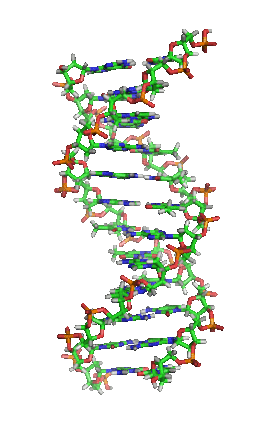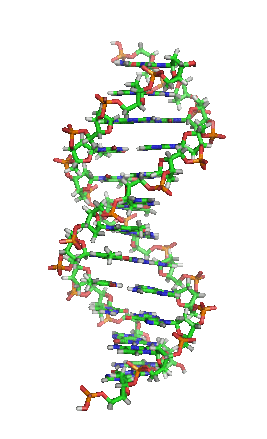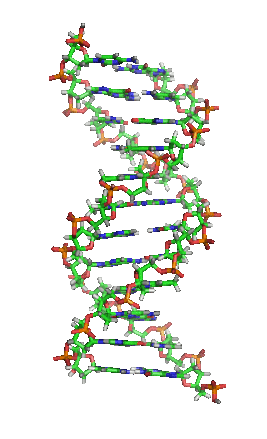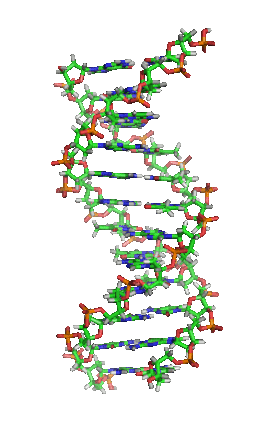
脱氧核糖核酸(英语:deoxyribonucleic acid,缩写:DNA)又称去氧核糖核酸,是一种生物大分子,可组成遗传指令,引导生物发育与生命机能运作。主要功能是信息储存,可比喻为“蓝图”或“配方”[1]。其中包含的指令,是建构细胞内其他的化合物,如蛋白质与核糖核酸所需。带有蛋白质编码的DNA片段称为基因。其他的DNA序列,有些直接以本身构造发挥作用,有些则参与调控遗传信息的表现。
DNA是一种长链聚合物,组成单位称为核苷酸,而糖类与磷酸借由酯键相连,组成其长链骨架。每个糖单位都与四种碱基里的其中一种相接,这些碱基沿着DNA长链所排列而成的序列,可组成遗传密码,是蛋白质氨基酸序列合成的依据。读取密码的过程称为转录,是根据DNA序列复制出一段称为RNA的核酸分子。多数RNA带有合成蛋白质的信息,另有一些本身就拥有特殊功能,例如核糖体RNA、小核RNA与小干扰RNA。
在细胞内,DNA能组织成染色体结构,整组染色体则统称为基因组。染色体在细胞分裂之前会先行复制,此过程称为DNA复制。对真核生物,如动物、植物及真菌而言,染色体是存放于细胞核内;对于原核生物而言,如细菌,则是存放在细胞质中的拟核里。染色体上的染色质蛋白,如组蛋白,能够将DNA组织并压缩,以帮助DNA与其他蛋白质进行交互作用,进而调节基因的转录。
一股脱氧核糖核酸上所具有的各类型含氮碱基,都只会与另一股上的一个特定类型碱基产生键结。此种情形称为互补性碱基配对。嘌呤与嘧啶之间会形成氢键,在一般情况下,A只与T相连,而C只与G相连。因此排列于双螺旋上的核苷酸,便以这种称为碱基对的方式相互联结。除此之外,与脱氧核糖核酸序列无关的疏水性效应,以及π重叠效应所产生的力,也是两股脱氧核糖核酸能维持结合状态的原因[2]。由于氢键比共价键更容易断裂,这使双股脱氧核糖核酸可能会因为机械力或高温作用,而有如拉链一般地解开[3],这种现象被称为DNA变性。由于互补的特性,使位于双股序列上的信息,皆以双倍的形式存在,这种特性对于脱氧核糖核酸复制过程来说相当重要。互补碱基之间可逆且具专一性的交互作用,是生物脱氧核糖核酸所共同拥有的关键功能[4]。
两种不同的碱基对分别是以不同数目的氢键结合:A-T之间有两条;G-C之间则有三条(如本段上方左图所示)。多一条氢键使GC配对的稳定性高于AT配对,也因此两股脱氧核糖核酸的结合强度,是由GC配对所占比例,以及双螺旋的总长度来决定。当脱氧核糖核酸双螺旋较长且GC含量较高时,其双股之间的结合能力较强;长度较短且AT含量较高时,结合能力则较弱[5]。双螺旋上有某些部位必须能够轻易解开,这些部位通常含有有较多的AT配对,例如细菌启动子上一段含有TATAAT序列的普里布诺盒[6]。在实验室中,若找出解开氢键所需的温度,也就是所谓熔点(Tm),便能计算出两股之间的结合强度。当脱氧核糖核酸双螺旋上所有的碱基配对都解开之后,溶液中的两股脱氧核糖核酸将分裂成独立的分子。单股脱氧核糖核酸分子并无固定的形体,但仍有某些形状较为稳定且常见[7]。
物理与化学性质
[编辑]
脱氧核糖核酸是一种由核苷酸重复排列组成的长链聚合物[4][8],宽度约22到24埃(2.2到2.4纳米),每一个核苷酸单位则大约长3.3埃(0.33纳米)[9]。在整个脱氧核糖核酸聚合物中,可能含有数百万个相连的核苷酸。例如人类细胞中最大的1号染色体中,就有2亿2千万个碱基对[10]。通常在生物体内,脱氧核糖核酸并非单一分子,而是形成两条互相配对并紧密结合[11][12],且如藤蔓般地缠绕成双螺旋结构的分子。每个核苷酸分子的其中一部分会相互连结,组成长链骨架;另一部分称为碱基,可使成对的两条脱氧核糖核酸相互结合。所谓核苷酸,是指一个核苷加上一个或多个磷酸基团,核苷则是指一个碱基加上一个糖类分子[13]。
脱氧核糖核酸骨架是由磷酸与糖类基团交互排列而成[14]。组成脱氧核糖核酸的糖类分子为环状的2-脱氧核糖,属于五碳糖的一种。磷酸基团上的两个氧原子分别接在五碳糖的3号及5号碳原子上,形成磷酸双酯键。这种两侧不对称的共价键位置,使每一条脱氧核糖核酸长链皆具方向性。双螺旋中的两股核苷酸互以相反方向排列,这种排列方式称为反平行。脱氧核糖核酸链上互不对称的两末端一边叫做5'端,另一边则称3'端。脱氧核糖核酸与RNA最主要的差异之一,在于组成糖分子的不同,DNA为2-脱氧核糖,RNA则为核糖[12]。
| 项目 | DNA | RNA | 解说 |
|---|---|---|---|
| 组成主干之糖类分子[12] | 2-脱氧核糖和磷酸 | 核糖和磷酸 | |
| 骨架结构 | 规则的[需要较佳来源][15]:50双螺旋结构[16] | 单螺旋结构[需要较佳来源][16] 或茎环结构[17] | 即脱氧核糖核酸由两条脱氧核苷酸链构成,而核糖核酸由一条核糖核苷酸链构成。[15]:49 |
| 核苷酸数 | 通常上百万 | 通常数百至数千个 | |
| 碱基种类[18][16] | 腺嘌呤(A)··· 胸腺嘧啶(T) 胞嘧啶(C)··· 鸟嘌呤(G) |
腺嘌呤(A)··· 尿嘧啶(U) 胞嘧啶(C)··· 鸟嘌呤(G) |
除部分例外,DNA为胸腺嘧啶(5-甲基尿嘧啶),RNA为尿嘧啶,使RNA更易被水解。 |
| 五碳糖种类[16] | 脱氧核糖 | 核糖 | |
| 五碳糖连接组成分 | 氢原子 | 羟基 | 在五碳糖的第二个碳原子上连接的组成分不同。 |
| 存在于(对于真核细胞而言)[16] | 细胞核(少量存在于线粒体、叶绿体) | 细胞质 |

两股脱氧核糖核酸长链上的碱基以氢键相互吸引,使双螺旋形态得以维持。这些碱基可分为两大类,以5角及6角杂环化合物组合而成的一类称为嘌呤;只有一个6角杂环的则称为嘧啶[13]。组成脱氧核糖核酸的碱基,分别是腺嘌呤(adenine,缩写A)、胞嘧啶(cytosine,C)、鸟嘌呤(guanine,G)与胸腺嘧啶(thymine,T)。碱基、糖类分子与磷酸三者结合之后,便成为完整的核苷酸。还有一种碱基称为尿嘧啶(uracil,U),此种碱基比胸腺嘧啶少了一个位于环上的甲基,一般出现在RNA分子中,角色相当于脱氧核糖核酸里的胸腺嘧啶。通常在脱氧核糖核酸中,它会作为胞嘧啶的分解产物,或是CpG岛中还未经甲基化的胞嘧啶突变产物。少见的例外发现于一种称为PBS1的细菌病毒,此类病毒的脱氧核糖核酸中含有尿嘧啶[19]。在某些特定RNA分子的合成过程中,会有许多尿嘧啶在酶的作用下失去一个甲基,因而转变成胸腺嘧啶,这种情形大多出现于一些在构造上具有功能,或者具有酶能力的RNA上,例如转运RNA与核糖体RNA[20]。
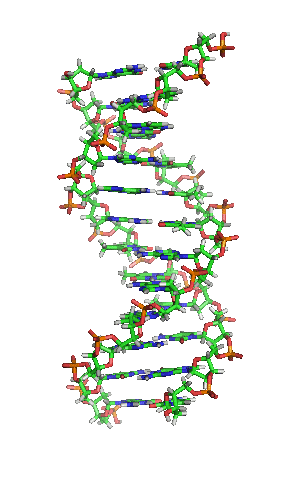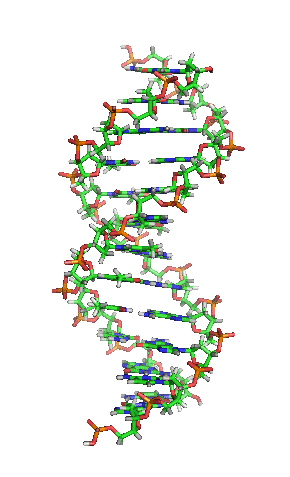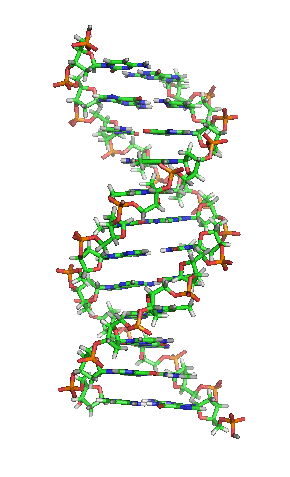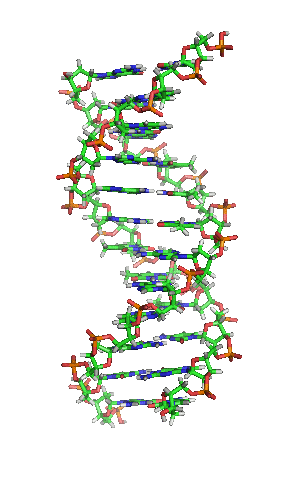
两股脱氧核糖核酸长链会以右旋方式交互缠绕成双螺旋结构,因为以磷酸联结而成的骨架位于外部,且两股之间会留下一些空隙,因此位于螺旋内部的碱基,即使从螺旋外侧依然可见(如右方动画)。双螺旋的表面有两种凹槽(或称“沟”):较大的宽22埃;较小的宽12埃[22]。由于各个碱基靠近大凹槽的一面较容易与外界接触,因此如转录因子等能够与特测序列结合的蛋白质与碱基接触时,通常是作用在靠近大凹槽的一面[23]。
碱基配对
[编辑]一股脱氧核糖核酸上所具有的各类型含氮碱基,都只会与另一股上的一个特定类型碱基产生键结。此种情形称为互补性碱基配对。嘌呤与嘧啶之间会形成氢键,在一般情况下,A只与T相连,而C只与G相连。因此排列于双螺旋上的核苷酸,便以这种称为碱基对的方式相互联结。除此之外,与脱氧核糖核酸序列无关的疏水性效应,以及π重叠效应所产生的力,也是两股脱氧核糖核酸能维持结合状态的原因[2]。由于氢键比共价键更容易断裂,这使双股脱氧核糖核酸可能会因为机械力或高温作用,而有如拉链一般地解开[3],这种现象被称为DNA变性。由于互补的特性,使位于双股序列上的信息,皆以双倍的形式存在,这种特性对于脱氧核糖核酸复制过程来说相当重要。互补碱基之间可逆且具专一性的交互作用,是生物脱氧核糖核酸所共同拥有的关键功能[4]。
两种不同的碱基对分别是以不同数目的氢键结合:A-T之间有两条;G-C之间则有三条(如本段上方左图所示)。多一条氢键使GC配对的稳定性高于AT配对,也因此两股脱氧核糖核酸的结合强度,是由GC配对所占比例,以及双螺旋的总长度来决定。当脱氧核糖核酸双螺旋较长且GC含量较高时,其双股之间的结合能力较强;长度较短且AT含量较高时,结合能力则较弱[5]。双螺旋上有某些部位必须能够轻易解开,这些部位通常含有有较多的AT配对,例如细菌启动子上一段含有TATAAT序列的普里布诺盒[6]。在实验室中,若找出解开氢键所需的温度,也就是所谓熔点(Tm),便能计算出两股之间的结合强度。当脱氧核糖核酸双螺旋上所有的碱基配对都解开之后,溶液中的两股脱氧核糖核酸将分裂成独立的分子。单股脱氧核糖核酸分子并无固定的形体,但仍有某些形状较为稳定且常见[7]。
正义与反义
[编辑]一般来说,当一段脱氧核糖核酸序列之mRNA为翻译成蛋白质的RNA序列时,称为“正义”(sense),而相对并互补的另一股序列,则称为“反义”(anti-sense)。由于RNA聚合酶的作用方式,是根据模板上的信息来合成一段与模板互补的RNA片段,因此正义mRNA的序列实际上与脱氧核糖核酸上的正义股相同。在同一股脱氧核糖核酸上,可能同时会有属于正义和反义的片段。此外,反义RNA在原核生物或真核生物体内皆存在,但是其功能尚未明了[24]。有研究认为,反义RNA可利用RNA与RNA之间的碱基配对,来调控基因的表现[25]。
少数属于原核生物、真核生物、质粒或病毒的脱氧核糖核酸序列(后两者较前两者多),会由于正义股与反义股之间的差异难以区分,而产生重叠基因[26],这类脱氧核糖核酸序列具有双重功能,一方面能以5'往3'的方向合成蛋白质,另一方面也能以相反方向合成另一个蛋白质。这种重叠现象一方面在细菌体内参与调控基因的转录[27],一方面则在较小的病毒基因组中,扮演增加信息量的角色[28]。为了缩减基因组的大小,也有某些病毒以线状或环状的单股脱氧核糖核酸作为遗传物质[29][30]。
超螺旋
[编辑]脱氧核糖核酸链在双螺旋基础上如绳索般扭转的现象与过程称为DNA超螺旋。当脱氧核糖核酸处于“松弛”状态时,双螺旋的两股通常会延着中轴,以每10.4个碱基对旋转一圈的方式扭转。但如果脱氧核糖核酸受到扭转,其两股的缠绕方式将变得更紧或更松[31]。当脱氧核糖核酸扭转方向与双股螺旋的旋转方向相同时,称为正超螺旋,此时碱基将更加紧密地结合。反之若扭转方向与双股螺旋相反,则称为负超螺旋,碱基之间的结合度会降低。自然界中大多数的脱氧核糖核酸,会因为拓扑异构酶的作用,而形成轻微的负超螺旋状态[32]。拓扑异构酶同时也在转录作用或DNA复制过程中,负责纾解脱氧核糖核酸链所受的扭转压强[33]。

各种类型的双螺旋结构
[编辑]脱氧核糖核酸有多种不同的构象,其中有些构象之间在构造上的差异并不大。目前已辨识出来的构象包括:A-DNA、B-DNA、C-DNA、D-DNA[34]、E-DNA[35]、H-DNA[36]、L-DNA[34]、P-DNA[37]与Z-DNA[14][38]。不过以现有的生物系统来说,自然界中可见的只有A-DNA、B-DNA与Z-DNA。脱氧核糖核酸所具有的构象可根据脱氧核糖核酸序列、超螺旋的程度与方向、碱基上的化学修饰,以及溶液状态,如金属离子与多胺浓度来分类[39]。三种主要构象中以B型为细胞中最常见的类型[40],与另两种脱氧核糖核酸双螺旋的差异,在于其几何形态与尺寸。
其中A型拥有较大的宽度与右旋结构,小凹槽较浅且较宽,大凹槽则较深较窄。A型一般存在于非生理状态的脱水样本中,在细胞中则可能为脱氧核糖核酸与RNA混合而成的产物(类似酶及脱氧核糖核酸的复合物)[41][42]。若一段脱氧核糖核酸上的碱基受到一种称为甲基化的化学修饰,将使其构型转变成Z型。此时螺旋形式转为左旋,与较常见的右旋B型相反[43]。某些专门与Z-脱氧核糖核酸结合的蛋白质可辨识出这种少见的结构,此外Z型脱氧核糖核酸可能参与了转录作用的调控[44]。
四联体结构
[编辑]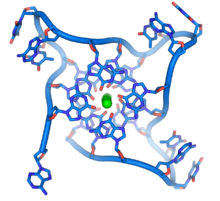
线状染色体的末端有一段称端粒的特殊区域,由于一般参与复制脱氧核糖核酸的酶无法作用于染色体的3'端,因此这些端粒的主要功能,是使细胞能利用一种称为端粒酶的酶来复制端粒[46]。如果端粒消失,那么复制过程将使染色体长度缩小。因此这些特化的端帽能保护染色体结尾不被外切酶破坏,并阻止细胞中的DNA修复系统将其视为需修正的损毁位置[47]。在人类细胞中,端粒是由重复出现数千次TTAGGG序列的单股脱氧核糖核酸所组成[48]。
这些序列富含鸟嘌呤,可形成一种由四个碱基重叠而成的特殊结构,使染色体末端较为稳定。四个鸟嘌呤可构成一个平面,并且重叠于其他平面之上,产生稳定的G-四联体结构[49]。碱基与位在四个碱基中心的金属离子螯合物之间,是经由氢键结合以稳定结构。左图显示由上方观看人类端粒中的四联体,图中可见每四个碱基为一组,共三层碱基重叠而成的单股脱氧核糖核酸环状物。在碱基环绕的中心,可见三个螯合在一起的钾离子[50]。也有其他类型的结构存在,例如中心的四个碱基,除了可以是属于单一的一股脱氧核糖核酸之外,也可能是由多条平行的脱氧核糖核酸各自贡献一个碱基而形成。
端粒另外还可形成一种大型环状结构,称为端粒环或T环(T-loop)。是由单股脱氧核糖核酸经过端粒结合蛋白的作用之后,卷曲而成的一个大循环[51]。在T环长链最前端的地方,单股的脱氧核糖核酸会附着在双股脱氧核糖核酸之上,破坏双螺旋脱氧核糖核酸与另一股的碱基配对,形成一种称为替代环或D环的三股结构[49]。
化学修饰
[编辑]碱基修饰
[编辑]基因的表现,受染色体上的染色质结构与异染色质(基因无表现或低表现)区域里的胞嘧啶甲基化所影响。举例而言,当胞嘧啶受到甲基化时,会转变成5-甲基胞嘧啶,此作用对于X染色体的去激活、铭印和保护脱氧核糖核酸分子不被内切酶所切断(存在例外)而言相当重要[52]。甲基化的程度在不同生物之间有所差异,如秀丽隐杆线虫便缺乏胞嘧啶甲基化,而在脊椎动物体内则较常出现,大约有1%的脱氧核糖核酸为5-甲基胞嘧啶[53]。5-甲基胞嘧啶容易因自然发生的脱氨作用而变成胸腺嘧啶,也因此使甲基化的胞嘧啶成为突变热点[54],这也解释了为什么胞嘧啶和鸟嘌呤会集中出现在CpG岛里,因为那里的甲基化作用被压制,没有甲基化的胞嘧啶所产生的突变产物并非胸腺嘧啶,而是尿嘧啶。因为尿嘧啶会相对容易地被更正过来,所以CpG岛内胞嘧啶不易形成突变而会被保留下来。其他的碱基修饰还包括细菌的腺嘌呤甲基化,以及使动质粒(一种生物)的尿嘧啶转变成“J-碱基”的糖基化等[55][56]。

脱氧核糖核酸损害
[编辑]有许多不同种类的突变原可对DNA造成损害,其中包括氧化剂、烷化剂,以及高频电磁辐射,如紫外线与X射线。不同的突变原对DNA造成不同类型的损害,举例而言,紫外线会造成胸腺嘧啶二聚体的形成,并与相邻的碱基产生交叉,进而使DNA发生损害[58]。另一方面,氧化剂如自由基或过氧化氢,可造成多种不同形态的损害,尤其可对鸟苷进行碱基修饰,并且使双股分解[59]。根据估计,在一个人类细胞中,每天大约有500个碱基遭受氧化损害[60][61]。在各种氧化损害当中,以双股分解最为危险,此种损害难以修复,且可造成DNA序列的点突变、插入与删除,以及染色体易位[62]。
许多突变原可嵌入相邻的两个碱基对之间,这些嵌入剂大多是芳香性分子及平面分子,包括乙锭、道诺霉素、阿霉素与沙利窦迈。必须先使碱基之间的空隙变大,才能使嵌入剂置入碱基对之间,整体而言,脱氧核糖核酸会因为双螺旋解开而扭曲变形。结构改变会使转录作用与脱氧核糖核酸复制过程受到抑制,进而导致毒害与突变。因此脱氧核糖核酸嵌入剂通常也是致癌物,常见的例子有二醇环氧苯并芘、吖啶、黄曲毒素与溴化乙锭等[63][64][65]。但是这些物质也因为能够抑制脱氧核糖核酸的转录与复制,而可应用于化学治疗中,用以抑制癌症细胞的快速生长情形[66]。
生物机能概观
[编辑]脱氧核糖核酸于真核生物细胞内,通常是以长条状染色体形式存在;在原核生物细胞内则是环状染色体。细胞内的所有染色体合称基因组。人类基因组中大约有30亿个碱基对,共组成了46个染色体[67]。脱氧核糖核酸所携带的信息,是以脱氧核糖核酸序列形式,保存于一些称为基因的片段中。基因中的遗传信息是经由互补的碱基配对来传递,例如在转录作用中,细胞里的RNA核苷酸会与互补的脱氧核糖核酸结合,复制出一段与脱氧核糖核酸序列互补的RNA序列。一般来说,这段RNA序列将会在翻译作用中,经由RNA之间的互补配对,合成出相对应的蛋白质序列。另一方面,细胞也可以在称为脱氧核糖核酸复制的过程中,单纯地复制其自身的遗传信息。
基因组结构
[编辑]真核生物的基因组脱氧核糖核酸主要存放于细胞核中,此外也有少量位于线粒体或叶绿体内。原核生物的脱氧核糖核酸则是保存在形状不规则的类核(nucleoid)当中[68]。基因是脱氧核糖核酸的一段区域,保存了基因组里的遗传信息,是遗传的单位,影响了生物个体的特定表征。基因中含有可转录的开放阅读框架,以及一些可调节开放阅读框架表现的调控序列,如启动子与增强子。
许多物种的基因组都只有一小部分可编译成蛋白质。以人类为例,在人类的基因组中只有1.5%属于含有蛋白质编码的外显子,另有超过50%属于无编码的重复序列[69]。真核生物基因组中如此大量的非编码DNA,以及物种之间不寻常的基因组大小或C值差异,长久以来一直是个难题,人们称之为“C值谜”[70]。不过这些不含蛋白质编码的脱氧核糖核酸序列,仍可能合成出具有功能的非编码RNA分子,用以调控基因表达[71]。
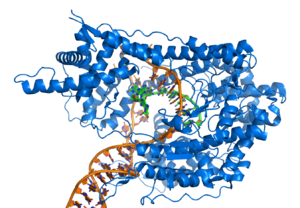
染色体中的某些非编码脱氧核糖核酸序列,本身具有结构上的功能。例如一般只带有少量基因的端粒与着丝粒,对于染色体的稳定性及机能而言显得相当重要[47][73]。人类体内有一类大量存在的非编码脱氧核糖核酸,称为伪基因,是一些因突变累积而变得残缺无用的基因复制品[74]。这些序列通常只可算是分子化石,不过有时候也会因为基因重复与趋异演化,而成为新基因里的新遗传物质[75]。
转录与翻译
[编辑]基因是指一段含有遗传信息,且可影响生物体表现型的脱氧核糖核酸序列。基因里的脱氧核糖核酸碱基序列决定了信使RNA的序列,而信使RNA的序列又决定了蛋白质的序列。翻译作用可依据基因所含有的核苷酸序列,以及遗传密码规则,生产出对应的蛋白质氨基酸序列。遗传密码的组成单位称为密码子,是含有三个字母的“指令”,这些单位则由三个核苷酸组成,例如ACT、CAG或TTT。
在转录作用中,基因里的密码子会在RNA聚合酶的作用下,复制成为信使RNA。之后核糖组会帮助带着氨基酸的转移RNA与信使RNA进行碱基配对,进而将信使RNA解码。由于组成密码子的碱基共有四种,且以三字母为一单位,因此可能存在的密码子一共有64种(43)。与这些密码子对应的标准氨基酸有20种,因此大多数氨基酸对应了一种以上的密码子。另外有三个密码子称为“终止密码子”或“无义密码子”,是编码区域的末端,分别是TAA、TGA与TAG,这是属于DNA上的终止密码。而在mRNA上的则是UAG、UAA与UGA。当翻译到达这三组密码子时就会停止翻译,并进行下一步的修饰。

复制
[编辑]生物个体成长需要经历细胞分裂,当细胞进行分裂时,必须将自身基因组中的脱氧核糖核酸复制,才能使子细胞拥有和亲代相同的遗传信息。脱氧核糖核酸的双股结构可供脱氧核糖核酸复制机制进行,在此复制过程中,两条长链会先分离,之后一种称为DNA聚合酶的酶,会分别以两条长链为依据,合成出互补的脱氧核糖核酸序列。酶可找出正确的外来互补碱基,并将其结合到模板长链上,进而制造出新的互补长链。由于脱氧核糖核酸聚合酶只能以5'到3'的方向合成脱氧核糖核酸链,因此双螺旋中平行但方向相反的两股,具有不同的合成机制[76]。旧长链上的碱基序列决定了新长链上的碱基序列,使细胞得以获得完整的脱氧核糖核酸复制品。
与蛋白质的交互作用
[编辑]脱氧核糖核酸若要发挥其功用,必须仰赖与蛋白质之间的交互作用,有些蛋白质的作用不具专一性,有些则只专门与个别的脱氧核糖核酸序列结合。聚合酶在各类酶中尤其重要,此种蛋白质可与脱氧核糖核酸结合,并作用于转录或脱氧核糖核酸复制过程。
脱氧核糖核酸结合蛋白
[编辑]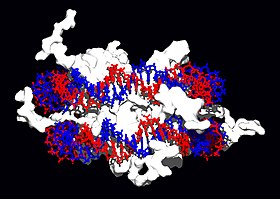
结构蛋白可与脱氧核糖核酸结合,是非专一性脱氧核糖核酸-蛋白质交互作用的常见例子。染色体中的结构蛋白与脱氧核糖核酸组合成复合物,使脱氧核糖核酸组织成紧密结实的染色质构造。对真核生物来说,染色质是由脱氧核糖核酸与一种称为组蛋白的小型碱性蛋白质所组合而成;而原核生物体内的此种结构,则掺杂了多种类型的蛋白质[77][78]。双股脱氧核糖核酸可在组蛋白的表面上附着并缠绕整整两圈,以形成一种称为核小体的盘状复合物。组蛋白里的碱性残基,与脱氧核糖核酸上的酸性糖磷酸骨架之间可形成离子键,使两者发生非专一性交互作用,也使复合物中的碱基序列相互分离[79]。在碱性氨基酸残基上所发生的化学修饰有甲基化、磷酸化与乙酰化等[80],这些化学作用可使脱氧核糖核酸与组蛋白之间的作用强度发生变化,进而使脱氧核糖核酸与转录因子接触的难易度改变,影响转录作用的速率[81]。其他位于染色体内的非专一性脱氧核糖核酸结合蛋白,还包括一种能优先与脱氧核糖核酸结合,并使其扭曲的高移动性群蛋白[82]。这类蛋白质可以改变核小体的排列方式,产生更复杂的染色质结构[83]。
脱氧核糖核酸结合蛋白中有一种专门与单股脱氧核糖核酸结合的类型,称为单股脱氧核糖核酸结合蛋白。人类的复制蛋白A是此类蛋白中获得较多研究的成员,作用于多数与解开双螺旋有关的过程,包括脱氧核糖核酸复制、重组以及脱氧核糖核酸修复[84]。这类结合蛋白可固定单股脱氧核糖核酸,使其变得较为稳定,以避免形成茎环(stem-loop),或是因为核酸酶的作用而水解。

相对而言,其他的蛋白质则只能与特定的脱氧核糖核酸序列进行专一性结合。大多数关于此类蛋白质的研究集中于各种可调控转录作用的转录因子。这类蛋白质中的每一种,都能与特定的脱氧核糖核酸序列结合,进而激活或抑制位于启动子附近序列的基因转录作用。转录因子有两种作用方式,第一种可以直接或经由其他中介蛋白质的作用,而与负责转录的RNA聚合酶结合,再使聚合酶与启动子结合,并开启转录作用[86]。第二种则与专门修饰组蛋白的酶结合于启动子上,使脱氧核糖核酸模板与聚合酶发生接触的难度改变[87]。
由于目标脱氧核糖核酸可能散布在生物体中的整个基因组中,因此改变一种转录因子的活性可能会影响许多基因的运作[88]。这些转录因子也因此经常成为信号传递过程中的作用目标,也就是作为细胞反映环境改变,或是进行分化和发育时的媒介。具专一性的转录因子会与脱氧核糖核酸发生交互作用,使脱氧核糖核酸碱基的周围产生许多接触点,让其他蛋白质得以“读取”这些脱氧核糖核酸序列。多数的碱基交互作用发生在大凹槽,也就是最容易从外界接触碱基的部位[89]。

脱氧核糖核酸修饰酶
[编辑]核酸酶与连接酶
[编辑]核酸酶是一种可经由催化磷酸双酯键的水解,而将脱氧核糖核酸链切断的酶。其中一种称为外切酶,可水解位于脱氧核糖核酸长链末端的核苷酸;另一种则是内切酶,作用于脱氧核糖核酸两个端点之间的位置。在分子生物学领域中使用频率最高的核酸酶为限制内切酶,可切割特定的脱氧核糖核酸序列。例如左图中的EcoRV可辨识出具有6个碱基的5′-GAT|ATC-3′序列,并从GAT与ATC之间那条垂直线所在的位置将其切断。此类酶在自然界中能消化噬菌体脱氧核糖核酸,以保护遭受噬菌体感染的细菌,此作用属于限制修饰系统的一部分[91]。在技术上,对序列具专一性的核酸酶可应用于分子选殖与脱氧核糖核酸指纹分析。
另一种酶脱氧核糖核酸连接酶,则可利用来自腺苷三磷酸或烟酰胺腺嘌呤二核苷酸的能量,将断裂的脱氧核糖核酸长链重新接合[92]。连接酶对于脱氧核糖核酸复制过程中产生的后随链而言尤其重要,这些位于复制叉上的短小片段,可在此酶作用下黏合成为脱氧核糖核酸模板的完整复制品。此外连接酶也参与了DNA修复与遗传重组作用[92]。
拓扑异构酶与螺旋酶
[编辑]拓扑异构酶是一种同时具有核酸酶与连接酶效用的酶,可改变脱氧核糖核酸的超螺旋程度。其中有些是先使脱氧核糖核酸双螺旋的其中一股切开以形成缺口,让另一股能穿过此缺口,进而减低超螺旋程度,最后再将切开的部位黏合[32]。其他类型则是将两股脱氧核糖核酸同时切开,使另一条双股脱氧核糖核酸得以通过此缺口,之后再将缺口黏合[93]。拓扑异构酶参与了许多脱氧核糖核酸相关作用,例如脱氧核糖核酸复制与转录[33]。
螺旋酶是分子马达的一种类型,可利用来自各种核苷三磷酸,尤其是腺苷三磷酸的化学能量,破坏碱基之间的氢键,使DNA双螺旋解开成单股形式[94]。此类酶参与了大多数关于DNA的作用,且必须接触碱基才能发挥功用。
聚合酶
[编辑]聚合酶是一种利用核苷三磷酸来合成聚合苷酸链的酶,方法是将一个核苷酸连接到另一个核苷酸的3'羟基位置,因此所有的聚合酶都是以5'往3'的方向进行合成作用[95]。在此类酶的激活位置上,核苷三磷酸分子会与单股聚合苷酸模板发生碱基配对,因而使聚合酶能够精确地依据模板,合成出互补的另一股聚合苷酸。聚合酶可依据所能利用的模板类型来做分类。
在脱氧核糖核酸复制过程中,依赖脱氧核糖核酸模板的DNA聚合酶可合成出脱氧核糖核酸序列的复制品。由于此复制过程的精确性是生命维持所必需,因此许多这类聚合酶拥有校正功能,可辨识出合成反应中偶然发生的配置错误,也就是一些无法与另一股配对的碱基。检测出错误之后,其3'到5'方向的外切酶活性会发生作用,并将错误的碱基移除[96]。大多数生物体内的脱氧核糖核酸聚合酶,是以称为复制体的大型复合物形式来发生作用,此复合物中含有许多附加的次单位,如DNA夹或螺旋酶[97]。
依赖RNA作为模板的脱氧核糖核酸聚合酶是一种较特别的聚合酶,可将RNA长链的序列复制成脱氧核糖核酸版本。其中包括一种称为逆转录酶的病毒酶,此种酶参与了逆转录病毒对细胞的感染过程;另外还有复制端粒所需的端粒酶[98][46],本身结构中含有RNA模板[47]。
转录作用是由依赖脱氧核糖核酸作为合成模板的RNA聚合酶来进行,此类酶可将脱氧核糖核酸长链上的序列复制成RNA版本。为了起始一个基因的转录,RNA聚合酶会先与一段称为启动子的脱氧核糖核酸序列结合,并使两股脱氧核糖核酸分离,再将基因序列复制成信使RNA,直到到达能使转录结束的终止子序列为止。如同人类体内依赖脱氧核糖核酸模板的脱氧核糖核酸聚合酶,负责转录人类基因组中大多数基因的RNA聚合酶II,也是大型蛋白质复合物的一部分,此复合物受到多重调控,也含有许多附加的次单位[99]。
遗传重组
[编辑]
各条脱氧核糖核酸螺旋间的交互作用不常发生,在人类细胞核里的每个染色体,各自拥有一块称作“染色体领域”的区域[101]。染色体之间在物理上的分离,对于维持脱氧核糖核酸信息储藏功能的稳定性而言相当重要。
不过染色体之间有时也会发生重组,在重组的过程中,会进行染色体互换:首先两条脱氧核糖核酸螺旋会先断裂,之后交换其片段,最后再重新黏合。重组作用使染色体得以互相交换遗传信息,并产生新的基因组合,进而增加自然选择的效果,且可能对蛋白质的演化产生重要影响[102]。遗传重组也参与脱氧核糖核酸修复作用,尤其是当细胞中的脱氧核糖核酸发生断裂的时候[103]。
同源重组是最常见的染色体互换方式,可发生于两条序列相类似的染色体上。而非同源重组则对细胞具有伤害性,会造成染色体易位与遗传异常。可催化重组反应的酶,如RAD51[104],称为“重组酶”。重组作用的第一个步骤,是内切酶作用,或是脱氧核糖核酸的损坏所造成的脱氧核糖核酸双股断裂[105]。重组酶可催化一系列步骤,使两条螺旋结合产生Holliday交叉。其中每条螺旋中的单股脱氧核糖核酸,皆与另一条螺旋上与之互补的脱氧核糖核酸连结在一起,进而形成一种可于染色体内移动的交叉形构造,造成脱氧核糖核酸链的互换。重组反应最后会因为交叉结构的断裂,以及脱氧核糖核酸的重新黏合而停止[106]。
脱氧核糖核酸生物代谢的演化
[编辑]脱氧核糖核酸所包含的遗传信息,是所有现代生命机能,以及生物生长与繁殖的基础。不过目前尚未明了在长达40亿年生命史中,脱氧核糖核酸究竟是何时出现并开始发生作用。有一些科学家认为,早期的生命形态有可能是以RNA作为遗传物质[107][108]。RNA可能在早期细胞代谢中扮演主要角色,一方面可传递遗传信息;另一方面也可作为核糖酶的一部分,进行催化作用[109]。在古代RNA世界里,核酸同时具有催化与遗传上的功能,而这些分子后来可能演化成为目前以四种核苷酸组成遗传密码的形式,这是因为当碱基种类较少时,复制的精确性会增加;而碱基种类较多时,增加的则是核酸的催化效能。两种可达成不同目的功能最后在四种碱基的情形下达到最合适数量[110]。
不过关于这种古代遗传系统并没有直接证据,且由于脱氧核糖核酸在环境中无法存留超过一百万年,在溶液中又会逐渐降解成短小的片段,因此大多数化石中并无脱氧核糖核酸可供研究[111]。即使如此,仍有一些声称表示已经获得更古老的DNA,其中一项研究表示,已从存活于2亿5千万年古老的盐类晶体中的细菌分离出脱氧核糖核酸[112],但此宣布引起了讨论与争议[113][114]。
技术应用
[编辑]遗传工程
[编辑]重组脱氧核糖核酸技术在现代生物学与生物化学中受到广泛应用,所谓重组DNA,是指集合其他脱氧核糖核酸序列所制成的人造脱氧核糖核酸,可以质粒或以病毒载体搭载所想要的格式,将脱氧核糖核酸转型到生物个体中[115]。经过遗传改造处里之后的生物体,可用来生产重组蛋白质,以供医学研究使用[116],或是于农业上栽种[117][118]。
法医鉴识
[编辑]法医可利用犯罪现场遗留的血液、精液、皮肤、唾液或毛发中的脱氧核糖核酸,来辨识可能的加害人。此过程称为遗传指纹分析或脱氧核糖核酸特征测定,此分析方法比较不同人类个体中许多的重复脱氧核糖核酸片段的长度,这些脱氧核糖核酸片段包括短串联重复序列与小卫星序列等,一般来说是最为可靠的罪犯辨识技术[119]。不过如果犯罪现场遭受多人的脱氧核糖核酸污染,那么将会变得较为复杂难解[120]。
首先于1984年发展脱氧核糖核酸特征测定的人是一名英国遗传学家阿莱克·杰弗里斯[121]。到了1988年,英国的谋杀案嫌犯科林·皮奇福克,成为第一位因脱氧核糖核酸特征测定证据而遭定罪者[122]。利用特定类型犯罪者的脱氧核糖核酸样本,可建立出数据库,帮助调查者解决一些只从现场采集到脱氧核糖核酸样本的旧案件。此外,脱氧核糖核酸特征测定也可用来辨识重大灾害中的罹难者[123]。
另外,有保险公司利用DNA鉴识技术,以确认理赔责任归属。相关事例包括:年仅16岁的台湾少年Lien-Yang Lee于2013年9月在澳洲遭遇车祸,他在申报保险理赔时声称,事故发生时他是坐后排乘客座上;不过,RACQ保险公司指驾驶座前安全气囊上血迹的DNA,是属于Lien-Yang Lee,故推断在车祸发生时他是坐在前排驾驶座上;故此,RACQ保险公司指他涉及诈欺,拒绝支付相关医疗费用;随后昆士兰州高等法院亦于2017年3月确认,Lien-Yang Lee在事故发生时涉及无照驾驶。[124]
历史学与人类学
[编辑]由于脱氧核糖核酸在经历一段时间后会积聚一些具有遗传能力突变,因此其中所包含的历史信息,可经由脱氧核糖核酸序列的比较,使遗传学家了解生物体的演化历史,也就是种系[125]。这些研究是系统发生学的一部分,也是演化生物学上的有利工具。假如对物种以内范围的脱氧核糖核酸序列进行比较,那么群体遗传学家就可得知特定种群的历史。此方法的应用范围可从生态遗传学到人类学,举例而言,脱氧核糖核酸证据已被试图用来寻找失踪的以色列十支派[126][127]。DNA也可以用来调查现代家族的亲戚关系,例如建构莎丽·海明斯与托马斯·杰斐逊的后代之间的家族关系,研究方式则与上述的犯罪调查相当类似,因此有时候某些犯罪调查案件之所以能解决,是因为犯罪现场的脱氧核糖核酸与犯罪者亲属的脱氧核糖核酸相符[128]。
生物信息学
[编辑]生物信息学影响了脱氧核糖核酸序列数据的运用、搜索与数据挖掘工作,并发展出各种用于储存并搜索脱氧核糖核酸序列的技术,可进一步应用于电脑科学,尤其是字串搜索算法、机器学习以及数据库理论[129]。字串搜索或比对算法是从较大的序列或较多的字母中,寻找单一序列或少数字母的出现位置,可发展用来搜索特定的核苷酸序列[130]。在其他如文本编辑器的应用里,通常可用简单的算法来解决问题,但只有少量可辨识特征的脱氧核糖核酸序列,却造成这些算法的运作不良。序列比对则试图辨识出同源序列,并定位出使这些序列产生差异的特定突变位置,其中的多重序列比对技术可用来研究种系发生关系及蛋白质的功能[131]。由整个基因组所构成的数据含有的大量脱氧核糖核酸序列,例如人类基因组计划的研究对象。若要将每个染色体上的每个基因,以及负责调控基因的位置都标示出来,会相当困难。脱氧核糖核酸序列上具有蛋白质或RNA编码特征的区域,可利用基因识别算法辨识出来,使研究者得以在进行实验以前,就预测出生物体内可能表现出来的特殊基因产物[132]。
脱氧核糖核酸与电脑
[编辑]
脱氧核糖核酸最早在运算上应用,是解决了一个属于NP完全的小型直接汉弥尔顿路径问题[133]。脱氧核糖核酸可作为“软件”,将信息写成核苷酸序列;并以酶或其他分子作为“硬件”进行读取或修饰。举例来说,作为硬件的限制酶FokI可以搭载一段具有软件功能的GGATG序列脱氧核糖核酸,再以其他的脱氧核糖核酸片段进行输入,并与软硬件复合物产生反应,最后输出另一段脱氧核糖核酸[134]。这种类似图灵机的装置可应用于药物治疗。此外脱氧核糖核酸运算在能源消耗、空间需求以及效率上优于电子电脑,且脱氧核糖核酸运算为具有高度平行(见平行运算)的计算方式。许多其他问题,包括多种抽象机器的模拟、布尔可满足性问题,以及有界形式的旅行推销员问题,皆曾利用脱氧核糖核酸运算做过分析[135]。由于小巧紧密的特性,脱氧核糖核酸也成为密码学理论的一部分,尤其在于能够利用脱氧核糖核酸有效地建构并使用无法破解的一次性密码本[136]。
脱氧核糖核酸与纳米科技
[编辑]脱氧核糖核酸的分子性质,例如自我组装特性,使其可用于某些纳米尺度的建构技术,例如利用脱氧核糖核酸作为模板,可导引半导体晶体的生长[137]。或是利用脱氧核糖核酸本身,来制成一些特殊结构,例如由脱氧核糖核酸长链交叉形成的脱氧核糖核酸“瓦片”(tile)[138]或是多面体[139]。此外也可以做出一些可活动的元件,例如纳米机械开关,此机械可经由使脱氧核糖核酸在不同的光学异构体(B型与Z型)之间进行转变,而使构形发生变化,导致开关的开启或关闭[140]。还有一种脱氧核糖核酸机械含有类似镊子的构造,可加入外来脱氧核糖核酸使镊子开合,并排出废物脱氧核糖核酸,此时脱氧核糖核酸的作用类似“燃料”[141]。脱氧核糖核酸所建构出来的装置,也可用来作为上述的脱氧核糖核酸运算工具。
历史
[编辑]
最早分离出脱氧核糖核酸的弗雷德里希·米歇尔是一名瑞士医生,他在1869年,从废弃绷带里所残留的脓液中,发现一些只有显微镜可观察的物质。由于这些物质位于细胞核中,因此米歇尔称之为“核素”(nuclein)[142]。到了1919年,菲巴斯·利文进一步辨识出组成脱氧核糖核酸的碱基、糖类以及磷酸核苷酸单元[143],他认为脱氧核糖核酸可能是许多核苷酸经由磷酸基团的联结,而串联在一起。不过他所提出概念中,脱氧核糖核酸长链较短,且其中的碱基是以固定顺序重复排列。1937年,威廉·阿斯特伯里完成了第一张X光衍射图,阐明了脱氧核糖核酸结构的规律性[144]。
1928年,弗雷德里克·格里菲斯从格里菲斯实验中发现,平滑型的肺炎球菌,能转变成为粗糙型的同种细菌,方法是将已死的平滑型与粗糙型活体混合在一起。这种现象称为“转型”。但造成此现象的因子,也就是脱氧核糖核酸,是直到1943年,才由奥斯瓦尔德·埃弗里等人所辨识出来[145]。1953年,阿弗雷德·赫希与玛莎·蔡斯确认了脱氧核糖核酸的遗传功能,他们在赫希-蔡斯实验中发现,脱氧核糖核酸是T2噬菌体的遗传物质[146]。
到了1953年,当时在卡文迪许实验室的詹姆斯·沃森与佛朗西斯·克里克,依据伦敦国王学院的罗莎琳·富兰克林所拍摄的X光衍射图[147]及相关数据,提出了[147]最早的核酸分子结构精确模型,并发表于《自然》期刊[11]。五篇关于此模型的实验证据论文,也同时以同一主题发表于《自然》[148]。其中包括富兰克林与雷蒙·葛斯林的论文[149],此文所附带的X光衍射图[150],是沃森与克里克阐明脱氧核糖核酸结构的关键证据。此外莫里斯·威尔金斯团队也是同期论文的发表者之一[151]。富兰克林与葛斯林随后又提出了A型与B型脱氧核糖核酸双螺旋结构之间的差异[152]。1962年,沃森、克里克以及威尔金斯共同获得了诺贝尔生理学或医学奖[153]。
克里克在1957年的一场演说中,提出了分子生物学的中心法则,预测了脱氧核糖核酸、RNA以及蛋白质之间的关系,并阐述了“转接子假说”(即后来的tRNA)[154]。1958年,马修·梅塞尔森与富兰克林·斯塔尔在梅塞尔森-斯塔尔实验中,确认了脱氧核糖核酸的复制机制[155]。后来克里克团队的研究显示,遗传密码是由三个碱基以不重复的方式所组成,称为密码子。这些密码子所构成的遗传密码,最后是由哈尔·葛宾·科拉纳、罗伯特·W·霍利以及马歇尔·沃伦·尼伦伯格解出[156]。这些发现代表了分子生物学的诞生。
为了测出所有人类的脱氧核糖核酸序列,人类基因组计划于1990年代展开。到了2001年,多国合作的国际团队与私人企业塞雷拉基因组公司,分别将人类基因组序列草图发表于《自然》[157]与《科学》[158]两份期刊。
参见
[编辑]参考文献
[编辑]- ^ Matt Ridley。蔡承志、许优优译。《23对染色体》(Genome)。商周出版。2000年。ISBN 978-957-667-678-9
- ^ 2.0 2.1 Ponnuswamy P, Gromiha M. On the conformational stability of oligonucleotide duplexes and tRNA molecules. J Theor Biol. 1994, 169 (4): 419 – 32. PMID 7526075.
- ^ 3.0 3.1 Clausen-Schaumann H, Rief M, Tolksdorf C, Gaub H. Mechanical stability of single DNA molecules. Biophys J. 2000, 78 (4): 1997 – 2007 [2007-06-20]. PMID 10733978. (原始内容存档 (PDF)于2019-09-24).
- ^ 4.0 4.1 4.2 Alberts, Bruce; Alexander Johnson; Julian Lewis; Martin Raff; Keith Roberts; Peter Walters. Molecular Biology of the Cell; Fourth Edition. New York and London: Garland Science. 2002 [2007-06-20]. ISBN 978-0-8153-3218-3. (原始内容存档于2009-10-03).
- ^ 5.0 5.1 Chalikian T, Völker J, Plum G, Breslauer K. A more unified picture for the thermodynamics of nucleic acid duplex melting: a characterization by calorimetric and volumetric techniques. Proc Natl Acad Sci U S A. 1999, 96 (14): 7853 – 8 [2007-06-20]. PMID 10393911. (原始内容存档 (PDF)于2019-09-24).
- ^ 6.0 6.1 deHaseth P, Helmann J. Open complex formation by Escherichia coli RNA polymerase: the mechanism of polymerase-induced strand separation of double helical DNA. Mol Microbiol. 1995, 16 (5): 817 – 24. PMID 7476180.
- ^ 7.0 7.1 Isaksson J, Acharya S, Barman J, Cheruku P, Chattopadhyaya J. Single-stranded adenine-rich DNA and RNA retain structural characteristics of their respective double-stranded conformations and show directional differences in stacking pattern. Biochemistry. 2004, 43 (51): 15996 – 6010. PMID 15609994.
- ^ Butler, John M. (2001) Forensic DNA Typing "Elsevier". pp. 14 – 15. ISBN 978-0-12-147951-0.
- ^ Mandelkern M, Elias J, Eden D, Crothers D. The dimensions of DNA in solution. J Mol Biol. 1981, 152 (1): 153 – 61. PMID 7338906.
- ^ Gregory S; et al. The DNA sequence and biological annotation of human chromosome 1. Nature. 2006, 441 (7091): 315 – 21. PMID 16710414.
- ^ 11.0 11.1 Watson J, Crick F. Molecular structure of nucleic acids; a structure for deoxyribose nucleic acid (PDF). Nature. 1953, 171 (4356): 737 – 8 [2007-06-20]. PMID 13054692. (原始内容存档 (PDF)于2014-08-23).
- ^ 12.0 12.1 12.2 Berg J., Tymoczko J. and Stryer L. (2002) Biochemistry. W. H. Freeman and Company ISBN 0-7167-4955-6
- ^ 13.0 13.1 Abbreviations and Symbols for Nucleic Acids, Polynucleotides and their Constituents (页面存档备份,存于互联网档案馆) IUPAC-IUB Commission on Biochemical Nomenclature (CBN) Accessed 03 Jan 2006
- ^ 14.0 14.1 Ghosh A, Bansal M. A glossary of DNA structures from A to Z. Acta Crystallogr D Biol Crystallogr. 2003, 59 (Pt 4): 620 – 6. PMID 12657780.
- ^ 15.0 15.1 薛金星. 中学教材全解 工具版 高中生物必修1 分子与细胞. 陕西人民教育出版社. ISBN 9787545015751.
- ^ 16.0 16.1 16.2 16.3 16.4 人民教育出版社. 生物1 必修 分子与细胞. ISBN 9787107176708.
- ^ Setubal, João Carlos. Meidanis, João. Introduction to computational molecular biology. 主编 聂理.《分子生物学导论》. World Pub. Corperation. 2003. ISBN 7-5062-6574-5. OCLC 880306796.
- ^ 生命的螺旋-DNA與RNA. 国立科学工艺博物馆.
- ^ Takahashi I, Marmur J. Replacement of thymidylic acid by deoxyuridylic acid in the deoxyribonucleic acid of a transducing phage for Bacillus subtilis. Nature. 1963, 197: 794 – 5. PMID 13980287.
- ^ Agris P. Decoding the genome: a modified view. Nucleic Acids Res. 2004, 32 (1): 223 – 38. PMID 14715921.[失效链接]
- ^ 来源:PDB 1D65 (页面存档备份,存于互联网档案馆)
- ^ Wing R, Drew H, Takano T, Broka C, Tanaka S, Itakura K, Dickerson R. Crystal structure analysis of a complete turn of B-DNA. Nature. 1980, 287 (5784): 755 – 8. PMID 7432492.
- ^ Pabo C, Sauer R. Protein-DNA recognition. Annu Rev Biochem: 293 – 321. PMID 6236744.
- ^ Hüttenhofer A, Schattner P, Polacek N. Non-coding RNAs: hope or hype?. Trends Genet. 2005, 21 (5): 289 – 97. PMID 15851066.
- ^ Munroe S. Diversity of antisense regulation in eukaryotes: multiple mechanisms, emerging patterns. J Cell Biochem. 2004, 93 (4): 664 – 71. PMID 15389973.
- ^ Makalowska I, Lin C, Makalowski W. Overlapping genes in vertebrate genomes. Comput Biol Chem. 2005, 29 (1): 1 – 12. PMID 15680581.
- ^ Johnson Z, Chisholm S. Properties of overlapping genes are conserved across microbial genomes. Genome Res. 2004, 14 (11): 2268 – 72. PMID 15520290.
- ^ Lamb R, Horvath C. Diversity of coding strategies in influenza viruses. Trends Genet. 1991, 7 (8): 261 – 6. PMID 1771674.
- ^ Davies J, Stanley J. Geminivirus genes and vectors. Trends Genet. 1989, 5 (3): 77 – 81. PMID 2660364.
- ^ Berns K. Parvovirus replication. Microbiol Rev. 1990, 54 (3): 316 – 29. PMID 2215424.
- ^ Benham C, Mielke S. DNA mechanics. Annu Rev Biomed Eng: 21 – 53. PMID 16004565.
- ^ 32.0 32.1 Champoux J. DNA topoisomerases: structure, function, and mechanism. Annu Rev Biochem: 369 – 413. PMID 11395412.
- ^ 33.0 33.1 Wang J. Cellular roles of DNA topoisomerases: a molecular perspective. Nat Rev Mol Cell Biol. 2002, 3 (6): 430 – 40. PMID 12042765.
- ^ 34.0 34.1 Hayashi G, Hagihara M, Nakatani K. Application of L-DNA as a molecular tag. Nucleic Acids Symp Ser (Oxf). 2005, 49: 261 – 262. PMID 17150733.
- ^ Vargason JM, Eichman BF, Ho PS. The extended and eccentric E-DNA structure induced by cytosine methylation or bromination. Nature Structural Biology. 2000, 7: 758 – 761. PMID 10966645.
- ^ Wang G, Vasquez KM. Non-B DNA structure-induced genetic instability. Mutat Res. 2006, 598 (1 – 2): 103 – 119. PMID 16516932.
- ^ Allemand; et al. Stretched and overwound DNA forms a Pauling-like structure with exposed bases. PNAS. 1998, 24: 14152–14157. PMID 9826669.
- ^ Palecek E. Local supercoil-stabilized DNA structures. Critical Reviews in Biochemistry and Molecular Biology. 1991, 26 (2): 151 – 226. PMID 1914495.
- ^ Basu H, Feuerstein B, Zarling D, Shafer R, Marton L. Recognition of Z-RNA and Z-DNA determinants by polyamines in solution: experimental and theoretical studies. J Biomol Struct Dyn. 1988, 6 (2): 299 – 309. PMID 2482766.
- ^ Leslie AG, Arnott S, Chandrasekaran R, Ratliff RL. Polymorphism of DNA double helices. J. Mol. Biol. 1980, 143 (1): 49–72. PMID 7441761.
- ^ Wahl M, Sundaralingam M. Crystal structures of A-DNA duplexes. Biopolymers. 1997, 44 (1): 45 – 63. PMID 9097733.
- ^ Lu XJ, Shakked Z, Olson WK. A-form conformational motifs in ligand-bound DNA structures. J. Mol. Biol. 2000, 300 (4): 819–40. PMID 10891271.
- ^ Rothenburg S, Koch-Nolte F, Haag F. DNA methylation and Z-DNA formation as mediators of quantitative differences in the expression of alleles. Immunol Rev: 286 – 98. PMID 12086319.
- ^ Oh D, Kim Y, Rich A. Z-DNA-binding proteins can act as potent effectors of gene expression in vivo. Proc. Natl. Acad. Sci. U.S.A. 2002, 99 (26): 16666–71. PMID 12486233.[失效链接]
- ^ 来自NDB UD0017 互联网档案馆的存档,存档日期2007-10-12.
- ^ 46.0 46.1 Greider C, Blackburn E. Identification of a specific telomere terminal transferase activity in Tetrahymena extracts. Cell. 1985, 43 (2 Pt 1): 405 – 13. PMID 3907856.
- ^ 47.0 47.1 47.2 Nugent C, Lundblad V. The telomerase reverse transcriptase: components and regulation. Genes Dev. 1998, 12 (8): 1073 – 85 [2007-06-20]. PMID 9553037. (原始内容存档于2007-09-27).
- ^ Wright W, Tesmer V, Huffman K, Levene S, Shay J. Normal human chromosomes have long G-rich telomeric overhangs at one end. Genes Dev. 1997, 11 (21): 2801 – 9 [2007-06-20]. PMID 9353250. (原始内容存档于2007-09-27).
- ^ 49.0 49.1 Burge S, Parkinson G, Hazel P, Todd A, Neidle S. Quadruplex DNA: sequence, topology and structure. Nucleic Acids Res. 2006, 34 (19): 5402 – 15 [2007-06-20]. PMID 17012276. (原始内容存档于2019-09-24).
- ^ Parkinson G, Lee M, Neidle S. Crystal structure of parallel quadruplexes from human telomeric DNA. Nature. 2002, 417 (6891): 876 – 80. PMID 12050675.
- ^ Griffith J, Comeau L, Rosenfield S, Stansel R, Bianchi A, Moss H, de Lange T. Mammalian telomeres end in a large duplex loop. Cell. 1999, 97 (4): 503 – 14. PMID 10338214.
- ^ Klose R, Bird A. Genomic DNA methylation: the mark and its mediators. Trends Biochem Sci. 2006, 31 (2): 89–97. PMID 16403636.
- ^ Bird A. DNA methylation patterns and epigenetic memory. Genes Dev. 2002, 16 (1): 6–21. PMID 11782440.
- ^ Walsh C, Xu G. Cytosine methylation and DNA repair. Curr Top Microbiol Immunol: 283–315. PMID 16570853.
- ^ Ratel D, Ravanat J, Berger F, Wion D. N6-methyladenine: the other methylated base of DNA. Bioessays. 2006, 28 (3): 309–15. PMID 16479578.
- ^ Gommers-Ampt J, Van Leeuwen F, de Beer A, Vliegenthart J, Dizdaroglu M, Kowalak J, Crain P, Borst P. beta-D-glucosyl-hydroxymethyluracil: a novel modified base present in the DNA of the parasitic protozoan T. brucei. Cell. 1993, 75 (6): 1129–36. PMID 8261512.
- ^ 来源:PDB 1JDG (页面存档备份,存于互联网档案馆)
- ^ Douki T, Reynaud-Angelin A, Cadet J, Sage E. Bipyrimidine photoproducts rather than oxidative lesions are the main type of DNA damage involved in the genotoxic effect of solar UVA radiation. Biochemistry. 2003, 42 (30): 9221–6. PMID 12885257.
- ^ Cadet J, Delatour T, Douki T, Gasparutto D, Pouget J, Ravanat J, Sauvaigo S. Hydroxyl radicals and DNA base damage. Mutat Res. 1999, 424 (1–2): 9–21. PMID 10064846.
- ^ Shigenaga M, Gimeno C, Ames B. Urinary 8-hydroxy-2′-deoxyguanosine as a biological marker of in vivo oxidative DNA damage (PDF). Proc Natl Acad Sci U S A. 1989, 86 (24): 9697–701 [2016-11-15]. PMID 2602371. (原始内容存档 (PDF)于2017-06-01).
- ^ Cathcart R, Schwiers E, Saul R, Ames B. Thymine glycol and thymidine glycol in human and rat urine: a possible assay for oxidative DNA damage (PDF). Proc Natl Acad Sci U S A. 1984, 81 (18): 5633–7 [2007-07-05]. PMID 6592579. (原始内容存档 (PDF)于2008-06-25).
- ^ Valerie K, Povirk L. Regulation and mechanisms of mammalian double-strand break repair. Oncogene. 2003, 22 (37): 5792–812. PMID 12947387.
- ^ Ferguson L, Denny W. The genetic toxicology of acridines. Mutat Res. 1991, 258 (2): 123–60. PMID 1881402.
- ^ Jeffrey A. DNA modification by chemical carcinogens. Pharmacol Ther. 1985, 28 (2): 237–72. PMID 3936066.
- ^ Stephens T, Bunde C, Fillmore B. Mechanism of action in thalidomide teratogenesis. Biochem Pharmacol. 2000, 59 (12): 1489–99. PMID 10799645.
- ^ Braña M, Cacho M, Gradillas A, de Pascual-Teresa B, Ramos A. Intercalators as anticancer drugs. Curr Pharm Des. 2001, 7 (17): 1745–80. PMID 11562309.
- ^ Venter J; et al. The sequence of the human genome. Science. 2001, 291 (5507): 1304–51. PMID 11181995.
- ^ Thanbichler M, Wang S, Shapiro L. The bacterial nucleoid: a highly organized and dynamic structure. J Cell Biochem. 2005, 96 (3): 506–21. PMID 15988757.
- ^ Wolfsberg T, McEntyre J, Schuler G. Guide to the draft human genome. Nature. 2001, 409 (6822): 824–6. PMID 11236998.
- ^ Gregory T. The C-value enigma in plants and animals: a review of parallels and an appeal for partnership. Ann Bot (Lond). 2005, 95 (1): 133–46 [2007-07-08]. PMID 15596463. (原始内容存档于2007-05-16).
- ^ The ENCODE Project Consortium. Identification and analysis of functional elements in 1% of the human genome by the ENCODE pilot project. Nature. 2007, 447 (7146): 799–816. ISSN 0028-0836. doi:10.1038/nature05874.
- ^ 来源:PDB 1MSW (页面存档备份,存于互联网档案馆)
- ^ Pidoux A, Allshire R. The role of heterochromatin in centromere function (PDF). Philos Trans R Soc Lond B Biol Sci. 2005, 360 (1455): 569–79. PMID 15905142.[失效链接]
- ^ Harrison P, Hegyi H, Balasubramanian S, Luscombe N, Bertone P, Echols N, Johnson T, 马克·本德·歌诗坦. Molecular fossils in the human genome: identification and analysis of the pseudogenes in chromosomes 21 and 22. Genome Res. 2002, 12 (2): 272–80 [2007-07-08]. PMID 11827946. (原始内容存档于2007-10-28).
- ^ Harrison P, 马克·本德·歌诗坦. Studying genomes through the aeons: protein families, pseudogenes and proteome evolution. J Mol Biol. 2002, 318 (5): 1155–74. PMID 12083509.
- ^ Albà M. Replicative DNA polymerases. Genome Biol. 2001, 2 (1): REVIEWS3002. PMID 11178285.[失效链接]
- ^ Sandman K, Pereira S, Reeve J. Diversity of prokaryotic chromosomal proteins and the origin of the nucleosome. Cell Mol Life Sci. 1998, 54 (12): 1350–64. PMID 9893710.
- ^ Dame RT. The role of nucleoid-associated proteins in the organization and compaction of bacterial chromatin. Mol. Microbiol. 2005, 56 (4): 858–70. PMID 15853876.
- ^ Luger K, Mäder A, Richmond R, Sargent D, Richmond T. Crystal structure of the nucleosome core particle at 2.8 A resolution. Nature. 1997, 389 (6648): 251–60. PMID 9305837.
- ^ Jenuwein T, Allis C. Translating the histone code. Science. 2001, 293 (5532): 1074–80. PMID 11498575.
- ^ Ito T. Nucleosome assembly and remodelling. Curr Top Microbiol Immunol: 1–22. PMID 12596902.
- ^ Thomas J. HMG1 and 2: architectural DNA-binding proteins. Biochem Soc Trans. 2001, 29 (Pt 4): 395–401. PMID 11497996.
- ^ Grosschedl R, Giese K, Pagel J. HMG domain proteins: architectural elements in the assembly of nucleoprotein structures. Trends Genet. 1994, 10 (3): 94–100. PMID 8178371.
- ^ Iftode C, Daniely Y, Borowiec J. Replication protein A (RPA): the eukaryotic SSB. Crit Rev Biochem Mol Biol. 1999, 34 (3): 141–80. PMID 10473346.
- ^ 来源:PDB 1LMB (页面存档备份,存于互联网档案馆)
- ^ Myers L, Kornberg R. Mediator of transcriptional regulation. Annu Rev Biochem: 729–49. PMID 10966474.
- ^ Spiegelman B, Heinrich R. Biological control through regulated transcriptional coactivators. Cell. 2004, 119 (2): 157–67. PMID 15479634.
- ^ Li Z, Van Calcar S, Qu C, Cavenee W, Zhang M, Ren B. A global transcriptional regulatory role for c-Myc in Burkitt's lymphoma cells. Proc Natl Acad Sci U S A. 2003, 100 (14): 8164–9 [2007-07-12]. PMID 12808131. (原始内容存档于2019-09-24).
- ^ Pabo C, Sauer R. Protein-DNA recognition. Annu Rev Biochem: 293–321. PMID 6236744.
- ^ Created from PDB 1RVA (页面存档备份,存于互联网档案馆)
- ^ Bickle T, Krüger D. Biology of DNA restriction. Microbiol Rev. 1993, 57 (2): 434–50. PMID 8336674.[失效链接]
- ^ 92.0 92.1 Doherty A, Suh S. Structural and mechanistic conservation in DNA ligases.. Nucleic Acids Res. 2000, 28 (21): 4051–8 [2007-07-12]. PMID 11058099. (原始内容存档于2019-09-24).
- ^ Schoeffler A, Berger J. Recent advances in understanding structure-function relationships in the type II topoisomerase mechanism. Biochem Soc Trans. 2005, 33 (Pt 6): 1465–70. PMID 16246147.
- ^ Tuteja N, Tuteja R. Unraveling DNA helicases. Motif, structure, mechanism and function. Eur J Biochem. 2004, 271 (10): 1849–63. PMID 15128295. doi:10.1111/j.1432-1033.2004.04094.x.
- ^ Joyce C, Steitz T. Polymerase structures and function: variations on a theme?. J Bacteriol. 1995, 177 (22): 6321–9 [2007-07-12]. PMID 7592405. (原始内容存档 (PDF)于2019-09-24).
- ^ Hubscher U, Maga G, Spadari S. Eukaryotic DNA polymerases. Annu Rev Biochem: 133–63. PMID 12045093.
- ^ Johnson A, O'Donnell M. Cellular DNA replicases: components and dynamics at the replication fork. Annu Rev Biochem: 283–315. PMID 15952889.
- ^ Tarrago-Litvak L, Andréola M, Nevinsky G, Sarih-Cottin L, Litvak S. The reverse transcriptase of HIV-1: from enzymology to therapeutic intervention. FASEB J. 1994, 8 (8): 497–503 [2007-07-12]. PMID 7514143. (原始内容存档于2008-09-05).
- ^ Martinez E. Multi-protein complexes in eukaryotic gene transcription. Plant Mol Biol. 2002, 50 (6): 925–47. PMID 12516863.
- ^ 来源:PDB 1M6G (页面存档备份,存于互联网档案馆)
- ^ Cremer T, Cremer C. Chromosome territories, nuclear architecture and gene regulation in mammalian cells. Nat Rev Genet. 2001, 2 (4): 292–301. PMID 11283701.
- ^ Pál C, Papp B, Lercher M. An integrated view of protein evolution. Nat Rev Genet. 2006, 7 (5): 337–48. PMID 16619049.
- ^ O'Driscoll M, Jeggo P. The role of double-strand break repair - insights from human genetics. Nat Rev Genet. 2006, 7 (1): 45–54. PMID 16369571.
- ^ Vispé S, Defais M. Mammalian Rad51 protein: a RecA homologue with pleiotropic functions. Biochimie. 1997, 79 (9-10): 587–92. PMID 9466696.
- ^ Neale MJ, Keeney S. Clarifying the mechanics of DNA strand exchange in meiotic recombination. Nature. 2006, 442 (7099): 153–8. PMID 16838012.
- ^ Dickman M, Ingleston S, Sedelnikova S, Rafferty J, Lloyd R, Grasby J, Hornby D. The RuvABC resolvasome. Eur J Biochem. 2002, 269 (22): 5492–501. PMID 12423347.
- ^ Joyce G. The antiquity of RNA-based evolution. Nature. 2002, 418 (6894): 214–21. PMID 12110897.
- ^ Orgel L. Prebiotic chemistry and the origin of the RNA world (PDF). Crit Rev Biochem Mol Biol: 99–123. PMID 15217990. (原始内容 (PDF)存档于2007-07-10).
- ^ Davenport R. Ribozymes. Making copies in the RNA world. Science. 2001, 292 (5520): 1278. PMID 11360970.
- ^ Szathmáry E. What is the optimum size for the genetic alphabet? (PDF). Proc Natl Acad Sci U S A. 1992, 89 (7): 2614–8 [2007-07-13]. PMID 1372984. (原始内容存档 (PDF)于2008-06-25).
- ^ Lindahl T. Instability and decay of the primary structure of DNA. Nature. 1993, 362 (6422): 709–15. PMID 8469282.
- ^ Vreeland R, Rosenzweig W, Powers D. Isolation of a 250 million-year-old halotolerant bacterium from a primary salt crystal. Nature. 2000, 407 (6806): 897–900. PMID 11057666.
- ^ Hebsgaard M, Phillips M, Willerslev E. Geologically ancient DNA: fact or artefact?. Trends Microbiol. 2005, 13 (5): 212–20. PMID 15866038.
- ^ Nickle D, Learn G, Rain M, Mullins J, Mittler J. Curiously modern DNA for a "250 million-year-old" bacterium. J Mol Evol. 2002, 54 (1): 134–7. PMID 11734907.
- ^ Goff SP, Berg P. Construction of hybrid viruses containing SV40 and lambda phage DNA segments and their propagation in cultured monkey cells. Cell. 1976, 9 (4 PT 2): 695–705. PMID 189942.
- ^ Houdebine L. Transgenic animal models in biomedical research. Methods Mol Biol: 163–202. PMID 17172731.
- ^ Daniell H, Dhingra A. Multigene engineering: dawn of an exciting new era in biotechnology. Curr Opin Biotechnol. 2002, 13 (2): 136–41. PMID 11950565.
- ^ Job D. Plant biotechnology in agriculture. Biochimie. 2002, 84 (11): 1105–10. PMID 12595138.
- ^ Collins A, Morton N. Likelihood ratios for DNA identification (PDF). Proc Natl Acad Sci U S A. 1994, 91 (13): 6007–11 [2007-07-15]. PMID 8016106. (原始内容存档 (PDF)于2007-11-27).
- ^ Weir B, Triggs C, Starling L, Stowell L, Walsh K, Buckleton J. Interpreting DNA mixtures. J Forensic Sci. 1997, 42 (2): 213–22. PMID 9068179.
- ^ Jeffreys A, Wilson V, Thein S. Individual-specific 'fingerprints' of human DNA.. Nature: 76–9. PMID 2989708.
- ^ Colin Pitchfork — first murder conviction on DNA evidence also clears the prime suspect 互联网档案馆的存档,存档日期2006-12-14. Forensic Science Service Accessed 23 Dec 2006
- ^ DNA Identification in Mass Fatality Incidents. National Institute of Justice. September 2006. (原始内容存档于2006-11-12).
- ^ Legal Frontline 法律前线通讯社. MAY 4 少年無照駕駛佯稱父親肇事 --- 血跡DNA揭謊言保險公司拒賠. [2017-08-14]. (原始内容存档于2017-08-14).
- ^ Wray G. Dating branches on the tree of life using DNA. Genome Biol. 2002, 3 (1): REVIEWS0001 [2007-07-15]. PMID 11806830. (原始内容存档于2019-09-24).
- ^ Lost Tribes of Israel, NOVA, PBS airdate: 22 February 2000. Transcript available from PBS.org, (页面存档备份,存于互联网档案馆) (last accessed on 4 March 2006)
- ^ Kleiman, Yaakov. "The Cohanim/DNA Connection: The fascinating story of how DNA studies confirm an ancient biblical tradition". (页面存档备份,存于互联网档案馆) aish.com (January 13, 2000). Accessed 4 March 2006.
- ^ Bhattacharya, Shaoni. "Killer convicted thanks to relative's DNA". (页面存档备份,存于互联网档案馆) newscientist.com (20 April 2004). Accessed 22 Dec 06
- ^ Baldi, Pierre. Brunak, Soren. Bioinformatics: The Machine Learning Approach MIT Press (2001) ISBN 978-0-262-02506-5
- ^ Gusfield, Dan. Algorithms on Strings, Trees, and Sequences: Computer Science and Computational Biology. Cambridge University Press, 15 January 1997. ISBN 978-0-521-58519-4.
- ^ Sjölander K. Phylogenomic inference of protein molecular function: advances and challenges. Bioinformatics. 2004, 20 (2): 170–9 [2007-07-15]. PMID 14734307. (原始内容存档于2008-10-11).
- ^ Mount DM. Bioinformatics: Sequence and Genome Analysis 2. Cold Spring Harbor, NY: Cold Spring Harbor Laboratory Press. 2004. ISBN 0879697121.
- ^ Adleman L. Molecular computation of solutions to combinatorial problems. Science. 1994, 266 (5187): 1021–4. PMID 7973651.
- ^ Benenson Y, Paz-Elizur T, Adar R, Keinan E, Livneh Z, Shapiro E. Programmable and autonomous computingmachine made of biomolecules. Nature. 2001, 414: 430–434.
- ^ Parker J. Computing with DNA.. EMBO Rep. 2003, 4 (1): 7–10. PMID 12524509.[失效链接]
- ^ Ashish Gehani, Thomas LaBean and John Reif. DNA-Based Cryptography (页面存档备份,存于互联网档案馆). Proceedings of the 5th DIMACS Workshop on DNA Based Computers, Cambridge, MA, USA, 14–15 June 1999.
- ^ Coffer, J. L.; et al. Dictation of the shape of mesoscale semiconductor nanoparticle assemblies by plasmid DNA. Appl. Phys. Lett. 1996, 69: 3851–3853.
- ^ Strong M. Protein Nanomachines. PLoS Biology. 2004, 2: e73.
- ^ Chen J, Seeman NC. Synthesis from DNA of a molecule with the connectivity of a cube. Nature. 1991, 350: 631–633.
- ^ Mao C, Sun W, Shen Z, Seeman NC. A nanomechanical device based on the B-Z transition of DNA. Nature. 1999, 397: 144–146.
- ^ Bernard Yurke, Andrew J. Turberfield, Allen P. Mills, Jr, Friedrich C. Simmel and Jennifer L. Neumann. A DNA-Fuelled Molecular Machine Made of DNA. Nature. 2000, 406: 605–608.
- ^ Dahm R. Friedrich Miescher and the discovery of DNA. Dev Biol. 2005, 278 (2): 274–88. PMID 15680349.
- ^ Levene P,. The structure of yeast nucleic acid. J Biol Chem. 1919, 40 (2): 415–24 [2007-07-17]. (原始内容存档于2009-06-29).
- ^ Astbury W,. Nucleic acid. Symp. SOC. Exp. Bbl. 1947, 1 (66).
- ^ Avery O, MacLeod C, McCarty M. Studies on the chemical nature of the substance inducing transformation of pneumococcal types. Inductions of transformation by a desoxyribonucleic acid fraction isolated from pneumococcus type III. J Exp Med. 1944, 79 (2): 137–158 [2007-07-17]. (原始内容存档于2007-09-27).
- ^ Hershey A, Chase M. Independent functions of viral protein and nucleic acid in growth of bacteriophage (PDF). J Gen Physiol. 1952, 36 (1): 39–56 [2007-07-17]. PMID 12981234. (原始内容存档 (PDF)于2008-10-01).
- ^ 147.0 147.1 Watson J.D. and Crick F.H.C. "A Structure for Deoxyribose Nucleic Acid". (页面存档备份,存于互联网档案馆) (PDF) Nature 171, 737–738 (1953). Accessed 13 Feb 2007.
- ^ Nature Archives Double Helix of DNA: 50 Years (页面存档备份,存于互联网档案馆)
- ^ Molecular Configuration in Sodium Thymonucleate. Franklin R. and Gosling R.G.Nature 171, 740–741 (1953)Nature Archives Full Text (PDF) (页面存档备份,存于互联网档案馆)
- ^ Crystallographic photo of Sodium Thymonucleate, Type B. "Photo 51." M…. 2012-05-25. (原始内容存档于2012-05-25).
- ^ Molecular Structure of Deoxypentose Nucleic Acids. Wilkins M.H.F., A.R. Stokes A.R. & Wilson, H.R. Nature 171, 738–740 (1953)Nature Archives (PDF) (页面存档备份,存于互联网档案馆)
- ^ Ev idence for 2-Chain Helix in Crystalline Structure of Sodium Deoxyribonucleate. Franklin R. and Gosling R.G. Nature 172, 156–157 (1953)Nature Archives, full text (PDF) (页面存档备份,存于互联网档案馆)
- ^ The Nobel Prize in Physiology or Medicine 1962 (页面存档备份,存于互联网档案馆) Nobelprize .org Accessed 22 Dec 06
- ^ Crick, F.H.C. On degenerate templates and the adaptor hypothesis (PDF). 互联网档案馆的存档,存档日期2008-10-01. genome.wellcome.ac.uk (Lecture, 1955). Accessed 22 Dec 2006
- ^ Meselson M, Stahl F. The replication of DNA in Escherichia coli. Proc Natl Acad Sci U S A. 1958, 44 (7): 671–82. PMID 16590258.
- ^ The Nobel Prize in Physiology or Medicine 1968 (页面存档备份,存于互联网档案馆) Nobelprize.org Accessed 22 Dec 06
- ^ International Human Genome Sequencing Consortium. Initial sequencing and analysis of the human genome. (PDF). Nature. 2001, 409: 860−921 [2007-07-25]. (原始内容存档 (PDF)于2007-07-12).
- ^ Venter, JC; et al. The sequence of the human genome. (PDF). Science. 2001, 291: 1304−1351 [2007-07-25]. (原始内容存档 (PDF)于2007-07-13).
延伸阅读
[编辑]- (中文) 林正焜、洪火树《认识DNA:下一波的医疗革命》。商周出版,2005年。ISBN 978-986-124-428-0
- (中文) Karl Drlica。周业仁译。《DNA的14堂课》(Understanding DNA and Gene Cloning)。天下文化,2002年。ISBN 978-986-417-077-7
- (中文) James D. Watson。陈正萱、张项译。《双螺旋─DNA结构发现者的青春告白》(The Double Helix)。时报出版,1998年。ISBN 978-957-13-2617-7
- (英文) Clayton, Julie. (Ed.). 50 Years of DNA, Palgrave MacMillan Press, 2003. ISBN 978-1-4039-1479-8
- (英文) Judson, Horace Freeland. The Eighth Day of Creation: Makers of the Revolution in Biology, Cold Spring Harbor Laboratory Press, 1996. ISBN 978-0-87969-478-4
- (英文) Olby, Robert. The Path to The Double Helix: Discovery of DNA, first published in October 1974 by MacMillan, with foreword by Francis Crick; ISBN 978-0-486-68117-7; the definitive DNA textbook, revised in 1994, with a 9 page postscript.
- (英文) Ridley, Matt. Francis Crick: Discoverer of the Genetic Code (Eminent Lives) HarperCollins Publishers; 192 pp, ISBN 978-0-06-082333-7 2006
- (英文) Rose, Steven. The Chemistry of Life, Penguin, ISBN 978-0-14-027273-4.
- (英文) Watson, James D. and Francis H.C. Crick. A structure for Deoxyribose Nucleic Acid (页面存档备份,存于互联网档案馆) (PDF). Nature 171, 737–738, 25 April 1953.
- (英文) Watson, James D. DNA: The Secret of Life ISBN 978-0-375-41546-3.
- (英文) Watson, James D. The Double Helix: A Personal Account of the Discovery of the Structure of DNA (Norton Critical Editions). ISBN 978-0-393-95075-5
- (英文) Watson, James D. "Avoid boring people and other lessons from a life in science" New York: Random House. ISBN 978-0-375-421844
- (英文) Calladine, Chris R.; Drew, Horace R.; Luisi, Ben F. and Travers, Andrew A. Understanding DNA, Elsevier Academic Press, 2003. ISBN 978-0-12-155089-9
外部链接
[编辑]- (中文) 核酸 (页面存档备份,存于互联网档案馆) - 生物化学教学网页
- (中文) 画说DNA (页面存档备份,存于互联网档案馆) - DNA from the beginning中文版,以动画介绍DNA
- (英文) DNA Interactive (页面存档备份,存于互联网档案馆) - Flash互动式介绍
- (英文) DNA from the beginning
- (英文) The Register of Francis Crick Personal Papers - 佛朗西斯·克里克的文章
- (英文) Double Helix 1953–2003 英国国家生物技术教育中心
- (英文) Double helix: 50 years of DNA (页面存档备份,存于互联网档案馆), Nature
- (英文) Rosalind Franklin's contributions to the study of DNA
- (英文) U.S. National DNA Day - DNA日
- (英文) Genetic Education Modules for Teachers (页面存档备份,存于互联网档案馆) - DNA from the Beginning学习指导
- (英文) Listen to Francis Crick and James Watson talking on the BBC in 1962, 1972, and 1974
- (英文) DNA under electron microscope (页面存档备份,存于互联网档案馆)
- (英文) DNA Articles - 论文与信息收集
- (英文) DNA coiling to form chromosomes
- (英文) DISPLAR: DNA binding site prediction on protein
- (英文) Dolan DNA Learning Center (页面存档备份,存于互联网档案馆)
- (英文) Olby, R. (2003) "Quiet debut for the double helix" Nature 421 (January 23): 402–405.
- (英文) Basic animated guide to DNA cloning (页面存档备份,存于互联网档案馆)
- (英文) DNA the Double Helix Game (页面存档备份,存于互联网档案馆) - 诺贝尔奖官方网站







